HiSeq X Ten 受託シーケンス
HiSeq X Ten ヒト全ゲノムシーケンス
HiSeq X Tenの特徴
- 圧倒的な総解析塩基数。
- ポリメラーゼによる伸長反応というスタンダードな解析原理。
- 大規模なヒト全ゲノムシーケンスをリーズナブルに実現。
HiSeq X Tenの原理
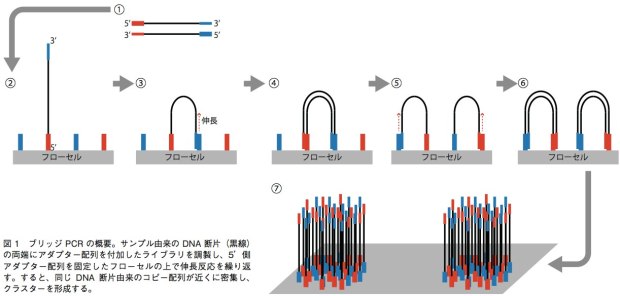
GenomeAnalyzer(GA)IIxやHiSeq、MiSeqでは、ブリッジPCRという方法を用いて、1本鎖DNA断片のクラスターをフローセル表面に作る。解析の際は、まずサンプルから得たDNA断片の両端に、2種類のアダプター配列(アダプター1、2)を連結させる。それを1本鎖にして、5’末端(アダプター1の側とする)をフローセル上に固定する。フローセル上には、あらかじめアダプター1、2と相補的に結合するプライマーが高密度に配置されており、サンプルの1本鎖DNAはアダプター2の側でこのプライマーと相補的に結合することになる(橋がかかったような構造になる)。この状態でDNAポリメラーゼによる伸長反応のあとで変性させると、フローセル上にはアダプター1側で結合した1本鎖と、アダプター2側で結合した1本鎖ができあがる。この反応を繰り返すことで、狭い面積の中で1本鎖DNAを固定しながら増幅することができる(図)。これを鋳型として、配列解析を行うのだ。塩基配列の解析には、蛍光標識したdNTPの取り込みを蛍光顕微鏡によって解析する。このdNTPは3’末端がブロックされており、1回の伸長反応で1塩基しか伸ばせない。そのため、1塩基ごとにどのdNTPが取り込まれたかを観察し、蛍光物質とブロックを外して次の伸長反応を行うというステップで、解析を進めていく。シングルリード法ではアダプター1の側からのみ、ペアエンド法ではアダプター1の側と2の側からそれぞれ最大で150bpを解析可能となっている。